La sang, com a òrgan líquid circulant, és relativament accessible com a mostra clínica. Entre les cèl·lules sanguínies, a banda dels tipus cel·lulars diferenciats, hi ha cèl·lules mare o troncals totipotencials. El seguiment dels diferents clons o llinatges d’aquestes cèl·lules desperta gran interès en possibles aplicacions diagnòstiques. D’entrada hom ha pensat d’identificar cada clon a partir de les variants genètiques que hagi incorporat en l’ADN a través de mutacions somàtiques. Ara bé, la taxa de mutació somàtica és massa baixa en condicions normals per aplicar aquesta estratègia. Tampoc no és gaire millor solució el seguiment a través d’eines d’enginyeria genètica. El Laboratori de Dinàmica Quantitativa de Cèl·lules Mare, d’Alejo Rodriguez-Fraticelli, i el Laboratori de Biologia Computacional i Genòmica de la Salut, de Lars Velten, han descobert que la metilació d’ADN d’un subconjunt de llocs CpG reflecteix la diferenciació cel·lular, mentre que la d’un altre subconjunt segueix epimutacions estocàstiques utilitzable com a codis de barres digitals de la identitat clonal. En un article a la revista Nature publicat ahir, amb Michael Scherer com a primer autor, demostren que el perfil de metilació d’ADN obtingut a partir d’una sola cèl·lula, si arriba a una resolució individual de cada CpG, pot oferir informació tant del grau de diferenciació com de l’origen clonal d’aquesta cèl·lula. Scherer et al. han desenvolupat EPI-Clone com a mètode per traçar llinatges cel·lulars sense recórrer a la transgènesi. L’han aplicat a l’hematopoiesi de ratolins i d’humans, i han estat capaços de rastrejar centenars de trajectòries de diferenciació clonal en desenes d’individus a partir de l’anàlisi de 230.358 cèl·lules individuals. Troben que el biaix mieloide que hom troba en l’hematopoiesi murina amb l’envelliment, així com la baixa producció de cèl·lules mare hematopoiètiques de ratolins vells, són restringides a un petit nombre de clons expandits, mentre que persisteixen fins a edat avançada molts clons funcionalment juvenils. En humans passa quelcom semblant.
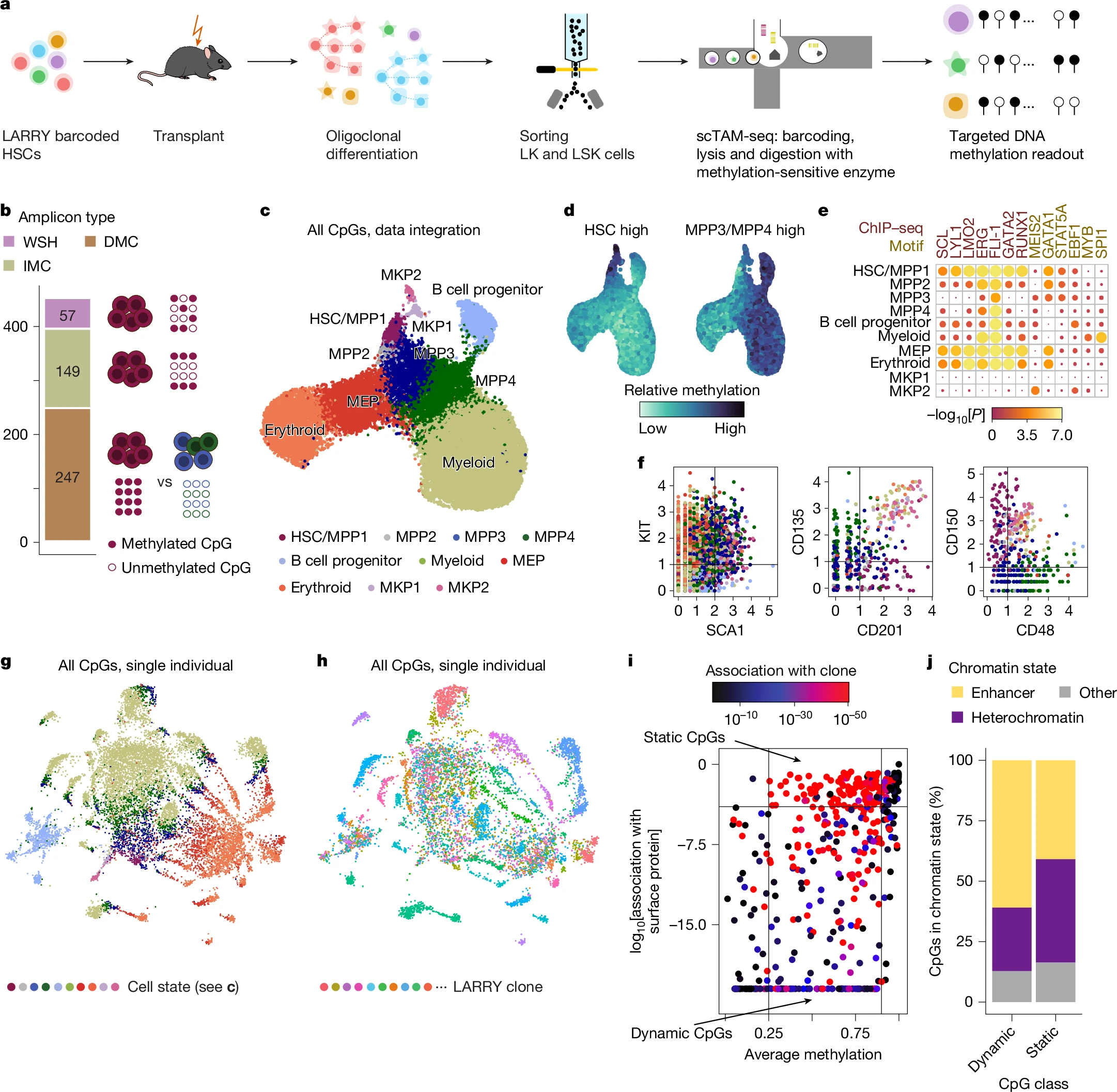
Scherer et al. mostren com la metilació d’ADN reflecteix alhora la diferenciació cel·lular i la identitat clonal. D’aquesta manera, epimutacions somàtiques en cèl·lules mare o troncals presents a la sang es poden fer servir com a marcadores de diferents clons o llinatges, i a través d’aquests hom pot fer un seguiment de l’envelliment sanguini
Traçar llinatges per entendre el desenvolupament i l’envelliment
Des de bon començament del segle XX l’embriologia comparada, i la biologia del desenvolupament que n’ha derivat, ha cercat formes de traçar els diferents llinatges cel·lulars que integren un cos. Els experimentadors han fet servir marcatges físics o genètics per distingir-los.
En termes generals, totes les cèl·lules d’un organisme comparteixen el mateix genoma, és a dir el mateix ADN. La diferència rau en la manera com s’expressa aquest genoma, és a dir en el transcriptoma, o conjunt de molècules d’ARN transcrites a partir de l’ADN. En els darrers anys s’ha fet possible una seqüenciació de tot l’ARN d’una sola cèl·lula (scRNA-seq), de manera que se’n pot extraure un codi de barres aplicable a la identificació dels clons derivats d’una cèl·lula troncal o a l’estat de cada cèl·lula. Aquesta metodologia, però, sol requerir eines complexes d’enginyeria genètiques, factibles en models experimentals, però no aplicables en humans.
L’alternativa passaria pel desenvolupament de marcadors clonals endògens, com ara són les mutacions somàtiques. La seqüenciació genòmica completa permet en aquest sentit la reconstrucció de filogènies cel·lulars en un mateix organisme, però en si mateix no ofereix informació sobre el grau i tipus de diferenciació.
L’ADN mitocondrial (mtDNA) és especialment interessant en aquest sentit perquè acumula mutacions somàtiques espontànies que poden seguir-se amb metodologies de scRNA-seq o d’ATAC-seq. Les variants de mtDNA ofereixen informació clonal i alhora sobre l’estat cel·lular.
Si la genètica molecular ens informa sobre mutacions en la seqüència d’ADN, l’epigenètica ho fa sobre modificadors que no alteren aquesta seqüència. Entre aquests modificadors destaca la metilació de l’ADN. El seguiment de la metilació d’ADN s’ha utilitzat per fer reconstruccions clonals en teixits sans i en neoplàsies. Hom pot parlar d’epimutacions somàtiques per referir-se a canvis espontanis però heretables en la metilació d’ADN. Aquestes epimutacions poden comportar una pèrdua de metilació d’ADN o bé un guany, i podrien ajudar a fer seguiments clonals en tumors. Ara bé, el procés de diferenciació cel·lular comporta en ell mateix canvis en la metilació d’ADN i això pot enfosquir les relacions clonals. Per evitar-ho cal disposar de mètodes de seqüenciació de la metilació d’ADN que ofereixin dades exhaustives de cada cèl·lula individual.
La sang és un òrgan excel·lent per provar aquestes idees. L’hematopoiesi, és a dir el procés pel qual es generen les cèl·lules sanguínies, ha estat estudiada de bell antuvi. Avui sabem que en l’ésser humà entre 50.000 i 200.000 clons de cèl·lules troncals participen en la generació de totes les cèl·lules sanguínies que tenim al llarg de la vida.
Amb l’envelliment es produeix una pèrdua de diversitat clonal, o el que és el mateix, alguns pocs clons s’expandeixen en detriment d’altres.
En ratolins hom ha seguit aquest comportament clonal en experiments de transplantaments de moll de l’os i en modelització matemàtica. En humans hom ha investigat les mutacions que promouen l’expansió clonal hematopoiètica, posant de manifest l’analogia amb els processos neoplàsics.
EPI-Clone és un mètode que explota la lectura de metilació d’ADN d’una cèl·lula fins a una resolució de CpG individual per traçar el seu origen clonal alhora que per extraure’n informació sobre el seu estat de diferenciació. L’anàlisi del metiloma (scTAM-seq) es fa en plataforma Mission Bio Tapestri i cobreix l’estat de metilació de centenars de CpGs en milers de cèl·lules, amb una taxa de fallida del 7%. scTAM-seq utilitza un enzim de restricció sensible a la metilació per difereix selectivament CpG no-metilades, la qual cosa li permet generar lectures de seqüència únicament de CpGs metilats.
Scherer et al. han aplicat EPI-Clone per a obtindre el codi de barres clonal de cèl·lules sanguínies humanes i murines. Han pogut caracteritzar el declivi en complexitat clonal associat a l’edat i quines són les propietats funcionals dels clons hematopoiètics que s’expandeixen en organismes vells.
El mapa de la metilació de l’ADN de l’hematopoiesi
Scherer et al. presenten vuit experiments de scTAM-seq fets en ratolí (M.1-M.8) i dotze mesures de scTAM-seq en dues cohorts humanes (A.1-A.7 i B.1-B.5). També presenten dos experiments que demostren la combinació de scTAM-seq amb RNA-seq i llinatge mitocondrial fets sobre la mateixa cèl·lula (X.1 i X.2).
Scherer et al. utilitzaren el sistema LARRY per marcar cèl·lules troncals hematopoiètiques (HSCs) de ratolí amb codis de barres lentivirals. Aquestes HSCs foren transplantades a ratolins que havien rebut un nivell letal d’irradiació. Al cap de cinc mesos feien un perfil de les poblacions cel·lulars sanguínies.
En l’experiment M.1 s’estudiaven cèl·lules troncals hematopoiètiques i progenitores (HSPCs) del moll de l’os d’aquests ratolins a través de scTAM-seq.
L’experiment M.2 era una rèplica de l’experiment M.1. En l’experiment M.3 la scTAM-seq s’aplicava en ratolins no-tractats, és a dir en una hematopoiesi nadiua.
En aquests experiments M.1-3 s’analitzava la metilació de 453 CpGs que havien seleccionats prèviament d’acord amb la bibliografia. En els experiments M.1-2, el codi de barres LARRY era llegit a partir d’un amplicó específic. En els experiments M.1-M.3 s’estudiava alhora l’expressió de 20 proteïnes de superfície amb anticossos marcats amb oligonucleòtids, i que fornia informació independent sobre diferenciació cel·lular.
Les dades de metilació dels experiments M.1-M.3 abasten 28.782 cèl·lules. Es pot seguir amb aquestes dades diferents trajectòries de diferenciació cel·lular com ara l’eritroide, la mieloide i la limfoide.
Per a anotar l’estat cel·lular en els experiments M.1-M.3, Scherer et al. consideren tres capes d’informació:
1) perfil global de metilació.
2) estat de metilació de llocs d’unió a factors de transcripció específics de llinatge (TFBSs).
3) expressió de les 20 proteïnes de superfície estudiades.
La desmetilació de CpGs properes a TFBSs es detecta per a GATA2 (un factor eritroide), EBF1 (un factor limfoide) i SPI1 (un factor mieloide). Les dades de scTAM-seq revelen l’existència d’un clúster de HSCs i de progenitors multipotents (MPP1, MPP2, MPP3 i MPP4), seguits de progenitors específics de les línies mieloide, eritroide, de limfòcits B i de megacariòcits.
La comparació amb scRNA-seq obtingudes d’altres cèl·lules però de les mateixes mostres dels experiments M.1-M.3 permet veure la relació entre transcriptoma i metiloma. La topologia és similar, i el transcriptoma reflecteix les quatre citades trajectòries de diferenciació.
La metilació d’ADN informa de l’origen clonal i de l’estat cel·lular
Les dades de l’experiment M.1 mostren com la metilació d’ADN captura dues capes d’informació: l’estat de diferenciació i la identitat clonal. L’associació de cada CpG amb l’expressió de proteïnes de superfície posa de manifest l’existència de CpGs dinàmics associats amb la diferenciació. Això mostra que la informació metilòmica és suficient per reconstruir l’estat de diferenciació de la cèl·lula. D’altra banda, els CpGs més estàtics són els que s’associen més amb la identitat clonal. Mentre que els CpGs dinàmics es troben enriquits en elements reguladors del genoma, els CpGs estàtics es localitzen preferencialment en regions heterocromàtiques o en dominis de replicació tardana.
L’algoritme EPI-Clone
Són, doncs, els CpGs estàtics els que reflectirien la identitat clonal. Scherer et al. han desenvolupat EPI-Clone en tres passes: 1) identificació dels CpGs estàtics; 2) identificació de cèl·lules de clons expandits; 3) caracterització de les cèl·lules de clons expandits.
EPI-Clone és capaç d’identificar cèl·lules que pertanyen a clons expandits en els experiments M.1-M.2 a través d’una anàlisi de components principals. Això indicaria que els senyals clonals epimutacionals es mantenen establement en els llinatges de cèl·lules sanguínies de ratolí durant un període de 5 mesos.
En l’experiment M.5 s’extreien cèl·lules immunitàries madures (limfoides i mieloides) del moll de l’os i de la melsa de ratolins. En aquestes cèl·lules s’obtenien dades d’expressió d’antígens de superfície i de metilació d’ADN en els CpGs ja esmentats. Els CpGs estàtics permetien trobar l’origen clonal d’aquestes cèl·lules immunitàries madures, especialment quan es tractaven de monòcits, neutròfils o limfòcits T CD8+. Era més difícil fer-ho en macròfags.
En l’experiment M.4 s’extreien cèl·lules mieloides madures del pulmó, del moll de l’os i de sang perifèrica de ratolins. La marca clonal de metilació d’ADN de CpGs estàtics es mantenia en la majoria de llinatges, excepte en macròfags.
En l’experiment M.6 s’extreien cèl·lules endotelials del pulmó d’un ratolí vell. El perfil metilòmic indica que en aquest llinatge cel·lular no-sanguini hi ha un conjunt similar de CpGs estàtics i dinàmics amb els quals es pot seguir, respectivament, l’origen clonal i l’estat de diferenciació.
Els clons hematopoiètics expandits en l’envelliment de ratolins
L’hematopoiesi és un procés policlonal en el sentit que diversos milers de clons contribueixen a la formació de cèl·lules sanguínies.
En l’experiment M.7 s’obtenien mostres de moll de l’os de dos ratolins joves de 12 setmanes d’edat. Vora el 50% de les cèl·lules obtingudes eren part de clons grans (amb contribucions individuals superiors a l’1%, i que en alguns casos arribaven al 4%). EPI-Clone és capaç d’identificar l’origen clonal de cèl·lules que pertanyen a aquests clons grans, però no pas als clons petits.
En l’experiment M.8, EPI-Clone era aplicat a ratolins joves de 12 setmanes i a ratolins vells de 100 setmanes. Entre les dues edats hi ha diferències en les proporcions de tipus cel·lulars. En ratolins vells els clons hematopoiètics són més expandits que en ratolins joves. Això implica una pèrdua de clonalitat amb l’edat.
En ratolins vells hi ha un augment moderat en el nombre de clons hematopoiètics que tenen un biaix mieloide.
EPI-Clone en moll de l’os humà
Per tal d’adaptar EPI-Clone a mostres humanes, Scherer et al. han seleccionat 448 CpGs amb metilació variable entre poblacions de progenitors sanguinis humans. També inclouen 147 regions genòmiques habitualment mutades en l’hematopoiesi i 20 regions del cromosoma Y.
Es colliren mostres de cèl·lules CD34+ de la sang de set donants A.1-A.7. Es va construir una base de dades de cèl·lules CD34+ de nou donants (B.1-B.5, X.1, A.1, A.3 i A.4) pels quals s’havien aconseguit més de 1000 cèl·lules de moll de l’os. En aquesta cohort hi havia identificades 10 mutacions hematopoiètiques i 1 cas de pèrdua de cromosoma Y. La caracterització fenotípica d’aquestes cèl·lules es feia amb un panell d’anticossos adreçat a 45 proteïnes de superfície.
En total s’obtingueren el perfil de scTAM-seq per a 135.432 cèl·lules individuals. EPI-Clone identifica un total de 67 expansions clonals en set donants.
Expansions clonals en envelliment humà
Dins dels clons hematopoiètics humans cal distingir entre els que presenten mutacions CH que afavoreixen l’expansió clonal i aquells que fan expansió clonal sense aquestes mutacions.
Els clons expandits en humans tendeixen a presentar un biaix en favor de la producció de cèl·lules mieloides en detriment de les línies de cèl·lula B i eritroides.
De les mostres X.1 s’obtingueren dades de metilació d’ADN i de RNA-seq per a 2745 cèl·lules. Els biaixos clonals s’associaven amb l’expressió de gens com TAL1, SLC40A1 i CDC45.
EPI-Clone i variants mitocondrials
La sang perifèrica del donant sa de 38 anys X.2 fou analitzada per EPI-Clone. Aquesta sang perifèrica ja havia estat analitzada per mt-scATAC-seq. En aquesta mostra foren identificats 44 clons hematopoiètics, amb expansions clonals de limfòcits NK i limfòcits T. De les 23 variants de mtDNA detectades, 5 tenien una relació filogenètica clara amb els clons hematopoiètics.
Aplicacions potencials d’EPI-Clone
EPI-Clone pot rastrejar el trànsit de la policlonalitat a l’oligoclonalitat que pateix l’hematopoiesi amb l’edat. Les dades d’aquest estudi indiquen que l’oligoclonalitat hematopoiètica comença vers els 50 anys d’edat, i es generalitza a 60 anys.
En aquesta oligoclonalitat no tan sols participen clons amb mutacions somàtiques CH sinó també clons sense mutacions conegudes. Els clons expandits amb mutacions CH mostren un major biaix cap a la generació mieloide, però no serien els únics en contribuir a la pèrdua de diversitat clonal i de capacitat hematopoiètica.
Lligams:
– Clonal tracing with somatic epimutations reveals dynamics of blood ageing. Michael Scherer, Indranil Singh, Martina Maria Braun, Chelsea Szu-Tu, Pedro Sanchez Sanchez, Dominik Lindenhofer, Niels Asger Jakobsen, Verena Körber, Michael Kardorff, Lena Nitsch, Pauline Kautz, Julia Rühle, Agostina Bianchi, Luca Cozzuto, Robert Frömel, Sergi Beneyto-Calabuig, Caleb Lareau, Ansuman T. Satpathy, Renée Beekman, Lars M. Steinmetz, Simon Raffel, Leif S. Ludwig, Paresh Vyas, Alejo Rodriguez-Fraticelli & Lars Velten. Nature (2025).
– “Codis de barres” escrits en el nostre ADN revelen com envelleix la sang, notícia a la pàgina web del CRG.